Публикация
Учени разработиха ефикасен, бърз и евтин тест за откриване на бактерии
Изследователи от Катедрата по физика на Норвежкия университет за наука и технологии (NTNU) и Института по физика на Китайската академия на науките в Пекин са разработили метод, който идентифицира бактериите лесно, евтино и по-точно от преди. Резултатите бяха представени в началото на септември в престижното списание Proceedings of the National Academy of Sciences (PNAS). Според учените това може да помогне за намаляване на употребата на антибиотици и ограничаване на антибиотичната резистентност.
„Разработихме прост инструмент, който може да идентифицира целия генетичен материал в бактериите. Това ни позволява да разберем по-бързо от какви бактерии е засегнат болен човек или животно или какви бактерии се намират в храната или околната среда. След идентификацията им можем да решим дали е необходимо да използваме антибиотици срещу бактерията и от какъв вид“, казва проф. Ерика Айзер.
Според учените една от причините, поради които новият метод е по-бърз, е, че
потребителите не трябва да преминават през стъпка
наречена „усилване на гена“, която включва създаване на няколко копия на генетичния материал, за да се анализира по-лесно.
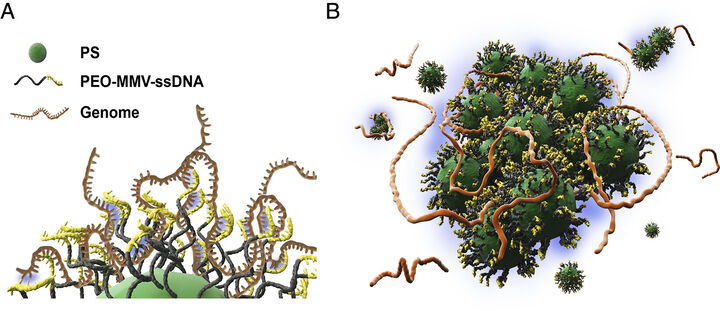
„Нашият подход използва ДНК-функционализирани полистиренови колоиди, които разграничават патогените въз основа на честотата на избрани къси ДНК последователности в техния геном. Важно е, че нашият метод използва цели геноми и не изисква амплификация на нуклеинова киселина. Полистиролови колоиди, присадени със специално проектирани повърхностни ДНК сонди, могат да се свързват съвместно с често повтарящи се последователности по целия геном на целевите бактерии, което води до образуването на големи и лесно откриваеми колоидни агрегати“, казват учените в своя абстракт към публикацията.
Те разказват, че използват полистиренови (PS) частици с микронни размери, присадени с 20-нуклеотидни (20-nt) дълги едноверижни ДНК (ssDNA) фрагменти, предназначени да хибридизират многовалентно с най-честите комплементарни последователности върху геномна ssDNA на мишената (в случая: Escherichia coli bl21-de3).
За да се провери
аналитичната надеждност и практическия потенциал на биосензора
за откриване на патогени в клинични проби, учените използват две проби с урина. Едната е чиста, а другата - с E. coli bl21-de3. Процедурата за екстракция на генома е извършена и върху двете проби, като тази, която е обогатена с целевия геном, показва много по-големи клъстери от тези, намерени в пробата от чиста урина.
„Откриваме, че имобилизирането на повърхностните ДНК проби върху PS колоиди води до агрегация на частици със силна флуоресценция при въвеждане на геномна ДНК на целевите бактерии. Тази флуоресценция може лесно да се наблюдава без предварителна PCR амплификация. ДНК-медиираният биосензор има няколко привлекателни характеристики, които го отличават от съществуващите биосензори. На първо място, след като аналитът се инжектира, за да реагира с ДНК-функционализирани PS частици, е необходимо по-малко от 1 минута за получаване на резултатите, което е значително по-бързо от съществуващите стратегии със сравнима чувствителност. Освен това, нашата система е в състояние да открие не-PCR-амплифицирани геноми в диапазон на концентрация на генома, обхващащ 10 порядъка“, отбелязват учените.
Те признават, че има известно ограничение във възможностите на прилаганата биосензорна техника. Отчита се, че подходът няма да работи за къси геноми, които са обичайни за вирусите. Според тях обаче този недостатък е предимство, тъй като това би означавало, че вирусните инфекции могат лесно да бъдат разграничени от бактериалните инфекции.
Цялото изследване можете да видите тук
По текста работи Бойко Бонев


Коментари